Que révèlent les microbes découverts sur les dessins de Léonard de Vinci ?
Le microbiote de sept dessins de Léonard de Vinci a été récemment étudié par une équipe interdisciplinaire. Que révèlent ces analyses ? Nous nous sommes entretenus avec Guadalupe Piñar pour en savoir plus.
Cette analyse a permis d’identifier des champignons, des bactéries et de l’ADN humain sur certains des dessins les plus célèbres de Léonard de Vinci, notamment Autoritratto et Uomo della Bitta. Une équipe composée d’historiens, de microbiologistes et de restaurateurs, dirigée par Guadalupe Piñar, a identifié les différents matériaux présents sur ces dessins.
Aujourd’hui, nous en apprenons plus sur ces découvertes avec Guadalupe Piñar, microbiologiste à l’Université des ressources naturelles et des sciences de la vie de Vienne.
Passéisme : Pouvez-vous nous parler de l’équipe de scientifiques derrière le projet et nous dire quel a été le déclencheur de cette étude ?
Guadalupe Piñar : L’équipe est interdisciplinaire, ce qui fait la particularité de cette étude qui ne serait pas possible sans le travail coordonné de tous les membres. L’équipe italienne est formée par M.C. Sclocchi et P. Colaizzi, tous deux scientifiques travaillant à l’ICPAL1 à Rome. Tous deux ont participé à l’échantillonnage et à l’analyse microscopique des échantillons. Maria Letizia Sebastiani est la directrice de l’ICPAL et a promu ce travail avec son intérêt et son financement. Flavia Pinzari est une ancienne scientifique de l’ICPAL, qui travaille maintenant à l’Institut des systèmes biologiques (IBS) du Conseil national italien de la recherche (CNR) et est une véritable professionnelle dans le domaine de la microscopie et de l’analyse MEB2.

L’équipe autrichienne est formée par Alexandra Graf, de l’unité de bio-informatique du campus FH de Vienne, qui a réalisé et analysé les données bio-informatiques. Katja Sterflinger travaille actuellement à l’Académie des Beaux-Arts de Vienne et est spécialiste de la microbiologie des matériaux du patrimoine culturel. Et moi-même, microbiologiste et biologiste moléculaire, qui a réalisé les analyses moléculaires de ce travail. L’expertise et le professionnalisme de tous les membres de l’équipe ont permis le succès de cette étude.
Son intérêt principal était de créer une bio-archive de chacun des dessins avec les informations biologiques contenues dans chacun d’eux au moment de l’échantillonnage. De cette manière, chaque bio-archive peut être utilisée comme un enregistrement pour des comparaisons futures, au cas où il pourrait y avoir des variations dans les conditions climatiques ou de stockage des dessins. Ces bio-archives sont également une valeur ajoutée pour les objets eux-mêmes qui sont en fait des dépositaires d’informations biologiques importantes.
Vous avez étudié la composition du matériel biologique présent dans sept dessins de Léonard de Vinci. Quelle méthode avez-vous utilisée et en quoi est-elle innovante ?
Nous avons utilisé une approche moléculaire, incluant le séquençage de toutes les informations biologiques contenues à la surface des dessins. La technologie de séquençage utilisée est l’une des plateformes de séquençage les plus innovantes actuellement disponibles, à savoir le séquençage Nanopore. Nanopore est une technologie qui sert à séquencer l’ADN (c’est-à-dire « lire » le code ADN contenu dans ses quatre bases ou « lettres » : ATGC). Nanopore est considéré comme une technologie de séquençage de troisième génération, qui offre plusieurs avantages par rapport aux technologies précédemment utilisées (technologies de séquençage NGS).
Les plates-formes NGS, bien que très utiles, présentent encore certaines limites, telles que le court longueur des fragments d’ADN qui peuvent être séquencés et les analyses bio-informatiques relativement complexes qui en découlent nécessaires au traitement des données obtenues. En outre, ils souffrent de certains biais dus à l’ACP3, ce qui est nécessaire dans ces techniques basées sur l’amplification clonale de l’ADN.

En revanche, Nanopore ne repose pas sur l’amplification de l’ADN clonal (comme les technologies de séquençage utilisées jusqu’à présent). Au lieu de cela, Nanopore séquence des molécules d’ADN « uniques » en temps réel alors qu’elles traversent un « nanopore » qui est intégré dans une membrane polymère électriquement résistante. Cette membrane a plusieurs de ces « nanopores » insérés, tous séquençant des molécules uniques d’ADN simultanément. La séquence d’ADN est ensuite lue sous l’effet d’un courant électrique.
Cela conduit à une simplicité de l’ensemble du processus de séquençage, d’abord par la réduction des réactifs et des produits chimiques (ce qui rend le processus moins cher et plus simple), et deuxièmement, en permettant une simplification des protocoles et des processus techniques en laboratoire (ce qui équivaut à un gain de temps).
De plus, cette technologie propose un appareil de détection de poche (plus petit qu’un téléphone portable), le MinION – celui utilisé dans cette étude -, qui offre les avantages d’un appareil portable. Donc, théoriquement, vous pouvez emporter l’appareil partout et faire le séquençage sur place (nous pouvons aussi imaginer pouvoir faire le séquençage dans des musées ou des archives, si nécessaire).
Est-ce la première fois qu’une telle méthode est appliquée à une œuvre d’art ou à un manuscrit ancien ?
Non, l’application pratique de cette technologie dans le domaine du patrimoine culturel a été lancée au cours des deux dernières années et est toujours en cours de développement, mais il existe peu d’études (cinq, à ce jour). Trois d’entre elles ont été appliquées par d’autres groupes de travail sur divers matériaux, tels que la cire, la pierre et les textiles. Nous avons publié deux études supplémentaires, la première appliquée sur la peinture sur toile et la seconde concernant les dessins de Léonard de Vinci.
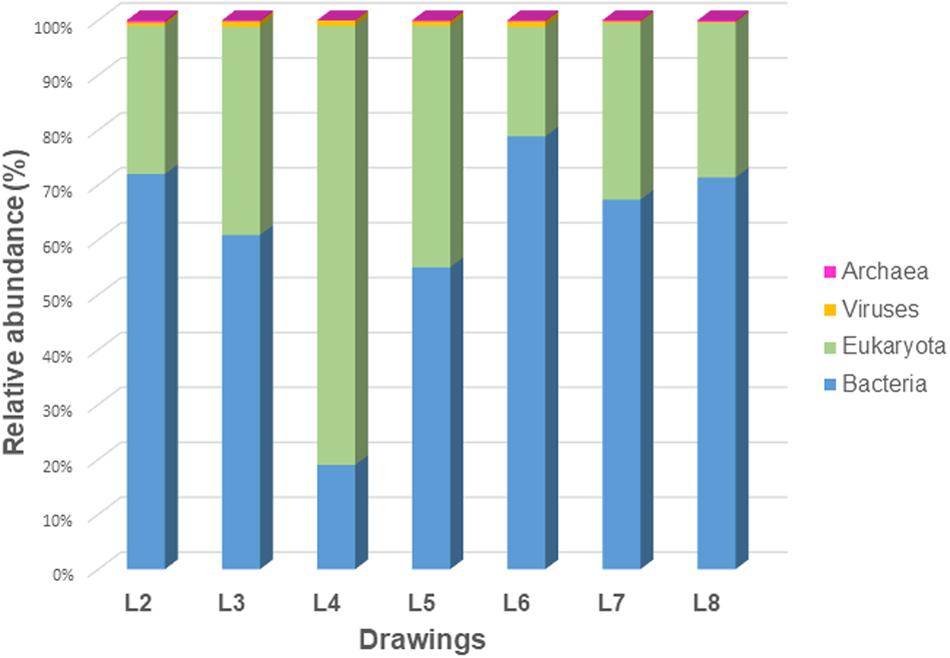
La différence de notre approche avec les approches suivies par les autres auteurs qui utilisent également Nanopore est qu’ils ont utilisé l’« amplification cible » axée sur le séquençage de régions spécifiques pour identifier les bactéries ou les champignons séparément. Dans nos études, la technologie de séquençage Nanopore a d’abord été appliquée en conjonction avec un protocole d’amplification du génome entier. Cette dernière stratégie nous a permis d’unifier les avantages que cette nouvelle technologie peut offrir et, en ne comptant pas sur l’amplification des régions cibles, nous pouvons montrer les proportions réelles de tous les domaines de vie présents dans les objets de valeur étudiés.
Une fois ces analyses effectuées, quelles sont les conclusions auxquelles vous êtes parvenus ?
Cette étude montre pour la première fois les microbiomes complets de certains des dessins les plus emblématiques de Léonard de Vinci. Cette information est une valeur ajoutée pour les dessins en termes de ce qu’ils peuvent révéler. En général, les informations biologiques contenues dans les dessins étudiés ont montré des microbiomes très spécifiques, qui peuvent être utilisés comme une bio-archive de l’histoire des objets, fournissant une sorte d’empreinte digitale pour les comparaisons actuelles et futures.
Cependant, certaines similitudes ont été observées qui pourraient être influencées par la situation géographique des dessins. Certains taxons n’ont été détectés que dans les dessins conservés à la Bibliothèque royale de Turin, tandis que d’autres taxons n’ont été détectés que dans les dessins conservés à la Bibliothèque Corsinian de Rome.

Les résultats ont montré une dominance surprenante des bactéries sur les champignons, ce qui contredit ce que l’on croyait sur les matériaux en papier. Ce fait est en partie dû à la stratégie d’échantillonnage utilisée, visant à prélever principalement les poussières déposées à la surface des dessins, mais reflète également la source de contamination à laquelle ils ont été soumis dans un passé récent, entraînant une forte proportion de bactéries typique du microbiome humain, introduit par une manipulation intensive des dessins lors des travaux de restauration, en plus d’autres micro-organismes qui ont manifestement été introduits comme contaminants par des vecteurs, tels que les insectes et leurs excréments.
Enfin, nous voulons souligner la valeur des échantillons pouvant être obtenus à partir d’œuvres d’art uniques. Des techniques analytiques basées sur l’étude de l’ADN environnemental permettront d’exploiter au maximum les bio-archives naturelles représentées par la poussière, le biofilm ou d’autres traces d’événements passés présents dans les œuvres. Dans ce souci, de nouvelles méthodes et protocoles sont nécessaires, tant pour la documentation que pour la récupération de ces « bio-archives » naturelles qui accompagnent tous les objets uniques de notre patrimoine artistique et culturel.
D’après vos recherches, l’ADN humain trouvé n’appartient pas au maître florentin, mais aux spécialistes qui ont restauré les dessins au fil du temps. Cette découverte aura-t-elle une conséquence sur la manière dont les objets à restaurer seront traités dans le futur ?
Honnêtement, nous n’avons jamais eu l’intention de faire une étude des séquences humaines dans ce travail. Les séquences d’ADN humain que nous avons trouvées n’ont pas été étudiées plus avant, mais interprétées comme une contamination par la manipulation des dessins au cours des siècles. Nous ne pouvons pas exclure complètement la présence de l’ADN du maître florentin, mais pour l’étudier, d’autres méthodes auraient été nécessaires et ce n’était pas le but de notre travail. Les résultats montrent une forte proportion d’ADN humain indiquant son origine plus récente, en particulier dans le dessin L4, ce qui indique une manipulation intense de ce dessin, très probablement lors des processus de restauration précédents.
Ce résultat indique que les techniques de restauration doivent être continuellement revues sur la base de preuves scientifiques et que les études de suivi dans le domaine de la conservation et de la restauration doivent être encouragées.
- L'ICPAL est l'Institut central pour la restauration et la conservation du patrimoine archivistique et du livre.
- MEB pour Microscopie électronique à balayage ou Scanning Electron Microscopy (SEM) en anglais.
- ACP pour Amplification en chaîne par polymérase ou Polymerase Chain Reaction (PCR) en anglais.



